Clonage de l’ADNc
L’ADNc est un segment monocaténaire d’ADN complémentaire de l’ARNm d’un segment d’ADN codant ou d’un gène entier. Il peut être utilisé comme sonde (sonde d’ADNc par opposition à une sonde génomique) pour le gène correspondant car il est complémentaire aux sections codantes (exons) du gène. Si le gène a été altéré par un réarrangement structural sur un site correspondant, par exemple par délétion, l’ADN normal et muté peuvent être différenciés. Ainsi, la préparation et le clonage de l’ADNc sont d’une grande importance. A partir de la séquence d’ADNc, des inférences essentielles peuvent être faites sur un gène et son produit génique. <fn>Watson, J.D., et al., Molecular Biology of the Gene, 3e éd. Benjamin / Cummings Publishing Co., Menlo Park, Californie, 1987. </fn>
Préparation de l’ADNc
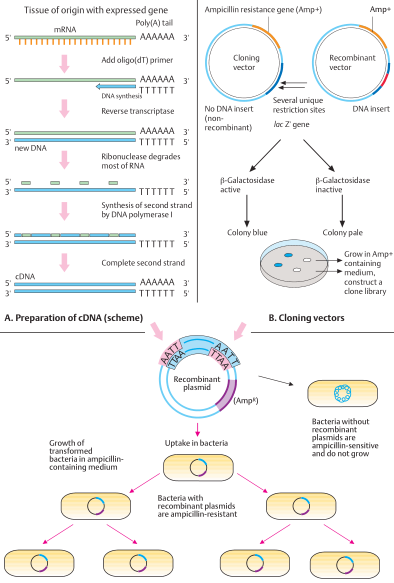
L’ADNc est préparé à partir de l’ARNm. Par conséquent, un tissu est requis dans lequel le gène respectif est transcrit et l’ARNm est produit en quantités suffisantes. Premièrement, l’ARNm est isolé. Ensuite, une amorce est attachée de sorte que l’enzyme transcriptase inverse puisse former de l’ADN complémentaire (ADNc) à partir de l’ARNm. Puisque l’ARNm contient du poly (A) à son extrémité 3 ‘, une amorce de poly (T) peut être attachée. A partir de là, l’enzyme transcriptase inverse peut commencer à former de l’ADNc dans la direction 5 ‘vers 3’. L’ARN est ensuite éliminé par la ribonucléase. L’ADNc sert de modèle pour la formation d’un nouveau brin d’ADN. Cela nécessite l’enzyme ADN polymérase. Le résultat est un double brin d’ADN, dont un brin est complémentaire de l’ARNm d’origine. A cet ADN, des séquences uniques (linkers) sont attachées qui sont complémentaires des extrémités monocaténaires produites par l’enzyme de restriction à utiliser. La même enzyme est utilisée pour couper le vecteur, par exemple un plasmide, de sorte que l’ADNc peut être incorporé pour le clonage.
Vecteurs de clonage
Le clonage cellulaire de fragments d’ADN de différentes tailles est facilité par une grande variété de systèmes de vecteurs. Les vecteurs plasmidiques sont utilisés pour cloner de petits fragments d’ADN dans des bactéries. Leur principal inconvénient est que seulement 5-10 kb d’ADN étranger peuvent être clonés. Un vecteur de clonage plasmidique qui a absorbé un fragment d’ADN (vecteur recombinant), par exemple pUC8 avec 2,7 kb d’ADN, doit être distingué de celui qui ne l’a pas. De plus, un gène de résistance à l’ampicilline (Amp +) sert à distinguer les bactéries qui ont absorbé des plasmides de celles qui n’en ont pas. Plusieurs sites de restriction uniques dans le segment d’ADN plasmidique où un fragment d’ADN pourrait être inséré servent de marqueurs avec un gène marqueur, tel que le gène lacZ. L’absorption d’un fragment d’ADN par un vecteur plasmidique perturbe le gène marqueur du plasmide. Ainsi, dans le plasmide recombinant, l’enzyme! la galactosidase ne sera pas produite par le gène lagZ rompu, alors que dans le plasmide sans insert d’ADN (non recombinant), l’enzyme est produite par le gène lacZ encore intact. L’activité du gène et la présence ou l’absence de l’enzyme sont déterminées en observant une différence de couleur des colonies en présence d’un substrat de sucre artificiel. Bêta-Glactosidase divise un sucre artificiel (5-bromo-4-chloro-3-indolyl! D -galactopyranoside) qui est similaire au lactose, le substrat naturel pour cette enzyme, en deux composants de sucre, dont l’un est bleu. Ainsi, les colonies bactériennes contenant des plasmides non recombinants avec un gène lacZ intact sont bleues. En revanche, les colonies qui ne sont pas des vecteurs recombinants restent pâles et blanches. Ces derniers sont cultivés dans un milieu contenant de l’ampicilline (le marqueur sélectionnable pour l’absorption des vecteurs plasmidiques). Par la suite, une bibliothèque de clones peut être construite. (Figure adaptée de Brown, 1999).
Clonage de l’ADNc
Seules ces bactéries deviennent résistantes à l’ampicilline et ont incorporé un plasmide recombinant. Les plasmides recombinants, qui contiennent le gène de résistance à l’ampicilline, transforment les bactéries sensibles à l’ampicilline en bactéries résistantes à l’ampicilline. Dans un milieu contenant de l’ampicilline, seules les bactéries qui contiennent le plasmide recombinant avec le fragment d’ADN désiré se développent. Par une réplication supplémentaire dans ces bactéries, le fragment peut être clone jusqu’à ce qu’il y ait suffisamment de matière à étudier. (Figures d’après Watson et al., 1987).
Principe du Clonage de l’ADNc
